Biomedizinische Wissensgraphen
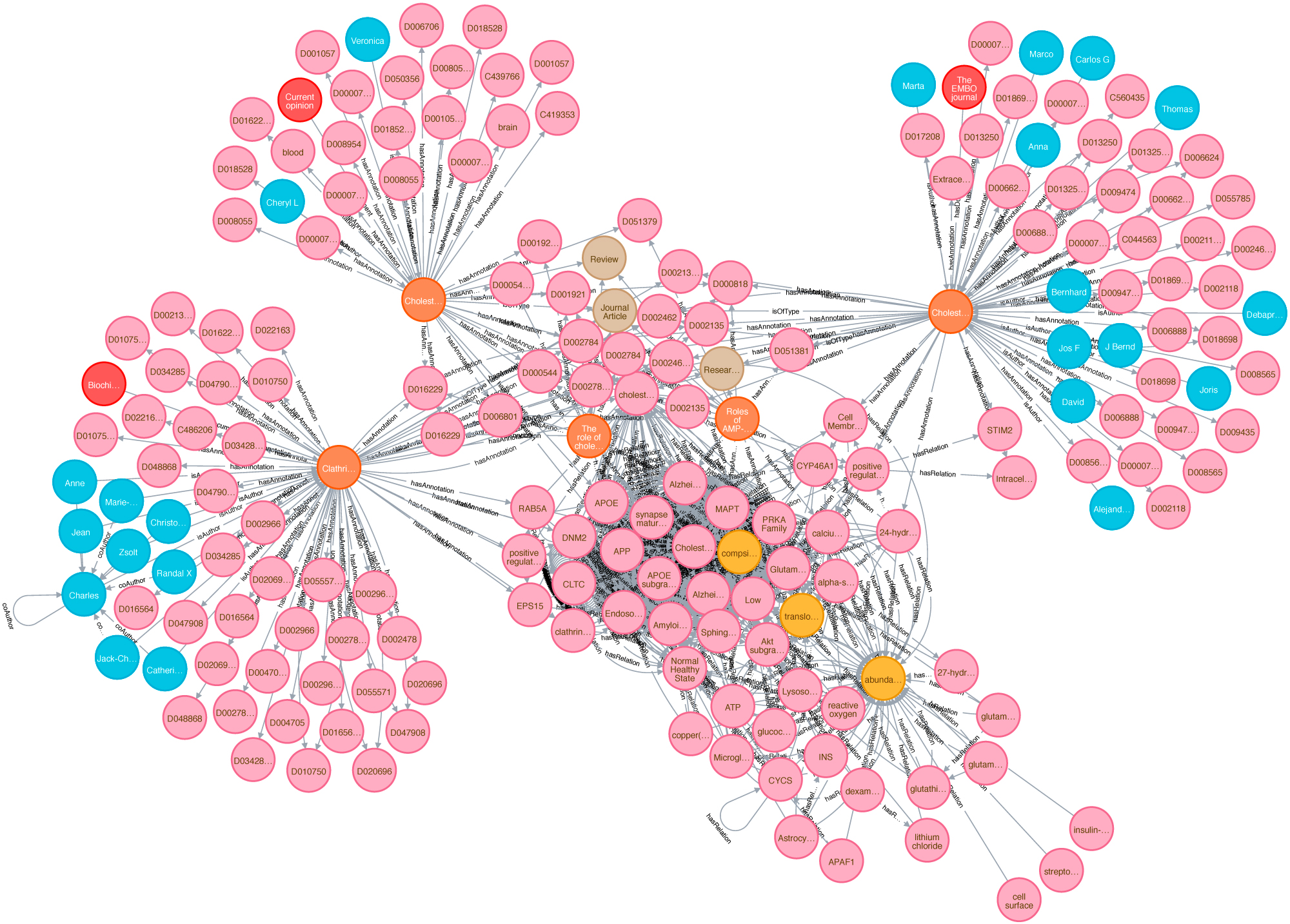
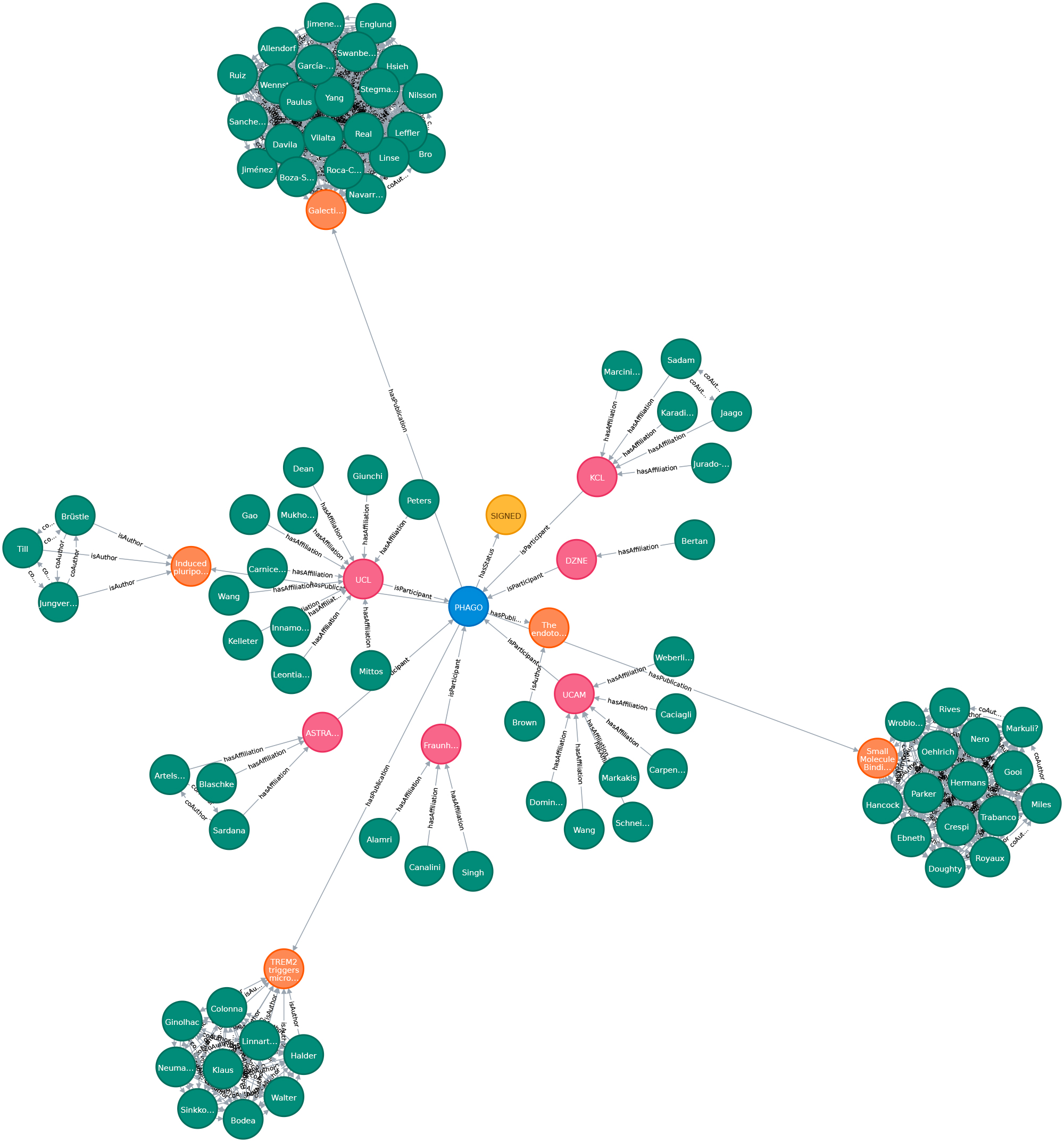
Biomedizinische Wissensgraphen spielen eine zentrale Rolle bei der Integration großer Datenmengen. Mit ihrer Hilfe lassen sich unstrukturierte Texte in ein strukturiertes, vergleichbares Format bringen. Als Ursache-Wirkungs-Modelle können Wissensgraphen potenziell die klinische Entscheidungsfindung erleichtern oder dazu beitragen, die Forschung in Richtung Präzisionsmedizin voranzutreiben. Daten- und Wissensmanagement, manchmal auch Informationsmanagement genannt, ist ein Kernthema von Data Science. Es ist auch ein interdisziplinäres Gebiet, das die Wirtschaftswissenschaften (wie effizient und teuer ist die Lösung?), die Psychologie (nutzen die Menschen diese Lösung in der beabsichtigten Weise?) und natürlich die Informatik tangiert. Unser Ziel ist der Aufbau einer nachhaltigen Dateninfrastruktur für biomedizinische Daten, personalisierte Medizin, Medikamentenneuverwendung, reproduzierbare KI und Wissensentdeckung.
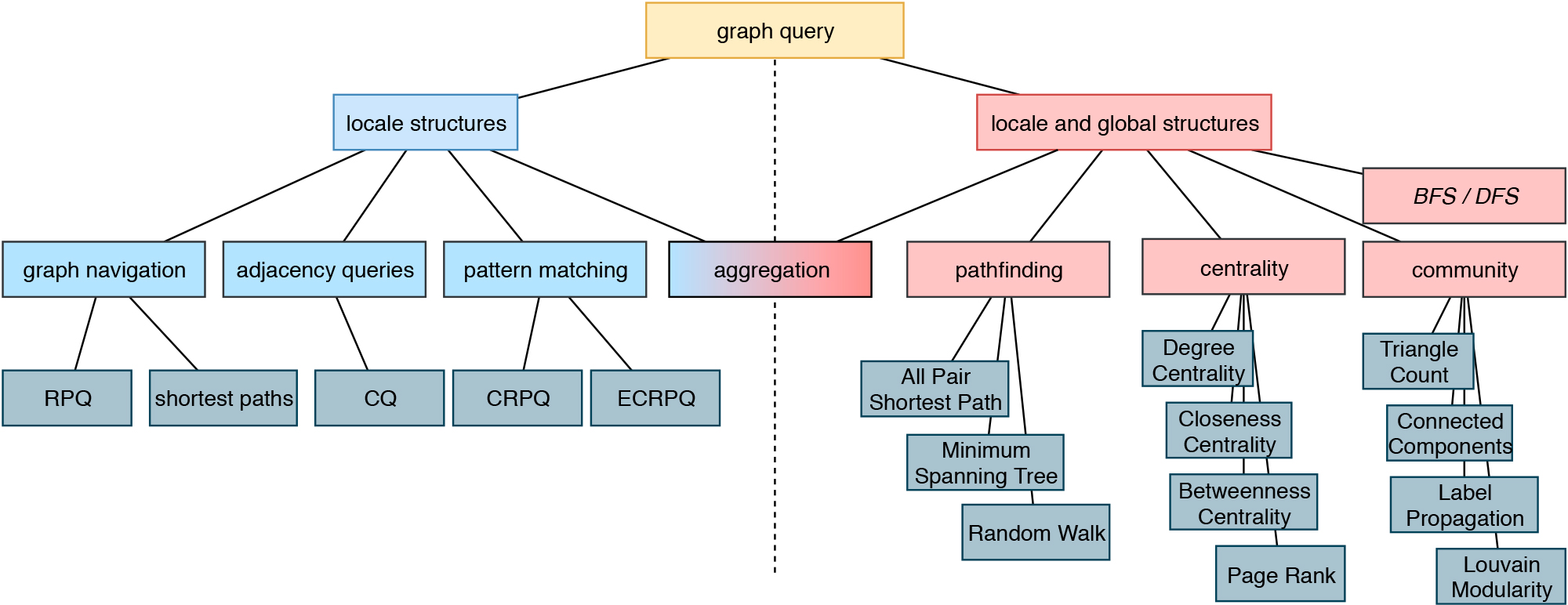
Ein »Wissensgraph« (manchmal auch als semantisches Netz bezeichnet) ist ein systematischer Weg, um Informationen und Datenpunkte mit Wissen zu verbinden. Es ist somit ein entscheidendes Konzept auf dem Weg zur Erzeugung von Wissen und Erkenntnis, zur Suche in Daten, Informationen und Wissen. Die Leistungsfähigkeit von Wissensgraphen hängt jedoch entscheidend von Kontextinformationen und Datenintegration ab. Hier bieten wir einen neuartigen semantischen Ansatz für einen kontextangereicherten biomedizinischen Wissensgraphen, indem wir die Datenintegration mit verknüpften Daten nutzen. Dieses Graphenkonzept kann für die Einbettung von Graphen verwendet werden, die in verschiedenen Ansätzen angewendet werden, z.B. mit Schwerpunkt auf der Themenerkennung und Wissensentdeckung. Daher ist die Verbindung von Wissensgraphen mit dem Kontext ein Schlüsselmerkmal. In diesem Projekt wollen wir einen neuartigen systematischen Ansatz zur Wissensentdeckung unter Verwendung von Kontexten in Wissensgraphen etablieren. Dazu reichern wir die bestehenden Graphenstrukturen an und bauen einen Kontext-Hypergraphen auf.
Wir erstellen einen riesigen Proof-of-Concept-Wissensgraphen mit beschrifteten Eigenschaftsgraphen, um Graphenalgorithmen zu testen und eine praktikable Umgebung zur Anwendung von semantischen Grapheneinbettungen zu schaffen. Es handelt sich dabei um eine hoch skalierbare, cloud-basierte Service-Umgebung. Dieses dichte, groß angelegte Testsystem verfügt derzeit über 75 Millionen Knoten und 960 Millionen Kanten. Die Grundlage für die Generierung unserer groß angelegten Wissensgraphendarstellung ist die biomedizinische Literatur (z.B. aus PubMed und PMC). Wir haben auch bibliographische Daten und Metadaten aus DBLP integriert, siehe hier. Da die aus SCAIView stammenden Basisdaten bereits mit verschiedenen biomedizinischen Ontologien annotiert sind, haben wir die CSO zu den DBLP-Daten annotiert. Wir haben unsere Grafik mit Daten aus dem Offenen Datenportal der EU (CORDIS - EU-Forschungsprojekte im Rahmen von Horizon 2020) angereichert. Dieser Datensatz kann sowohl für kommerzielle als auch für nicht-kommerzielle Zwecke wiederverwendet werden. Hier haben wir Projekte, ihren Status, Affiliationen, Personen und Autoren der in ihrem Datensatz erwähnten Publikationen integriert.
Download eines Ausschnitts des PHAGO Graphen
Dies ist die Grundlage für die Beantwortung von semantischen Fragen, Graphenabfragen und Erweiterungen auf der Basis von NLP, Text Mining, FAIR-Daten und ein Schritt in Richtung reproduzierbare KI. Dieser Graph ermöglicht den Vergleich von Forschungsdatensätzen aus verschiedenen Quellen sowie die Auswahl relevanter Datensätze mit Hilfe graphentheoretischer Algorithmen.
Medizinischer Wissensraum
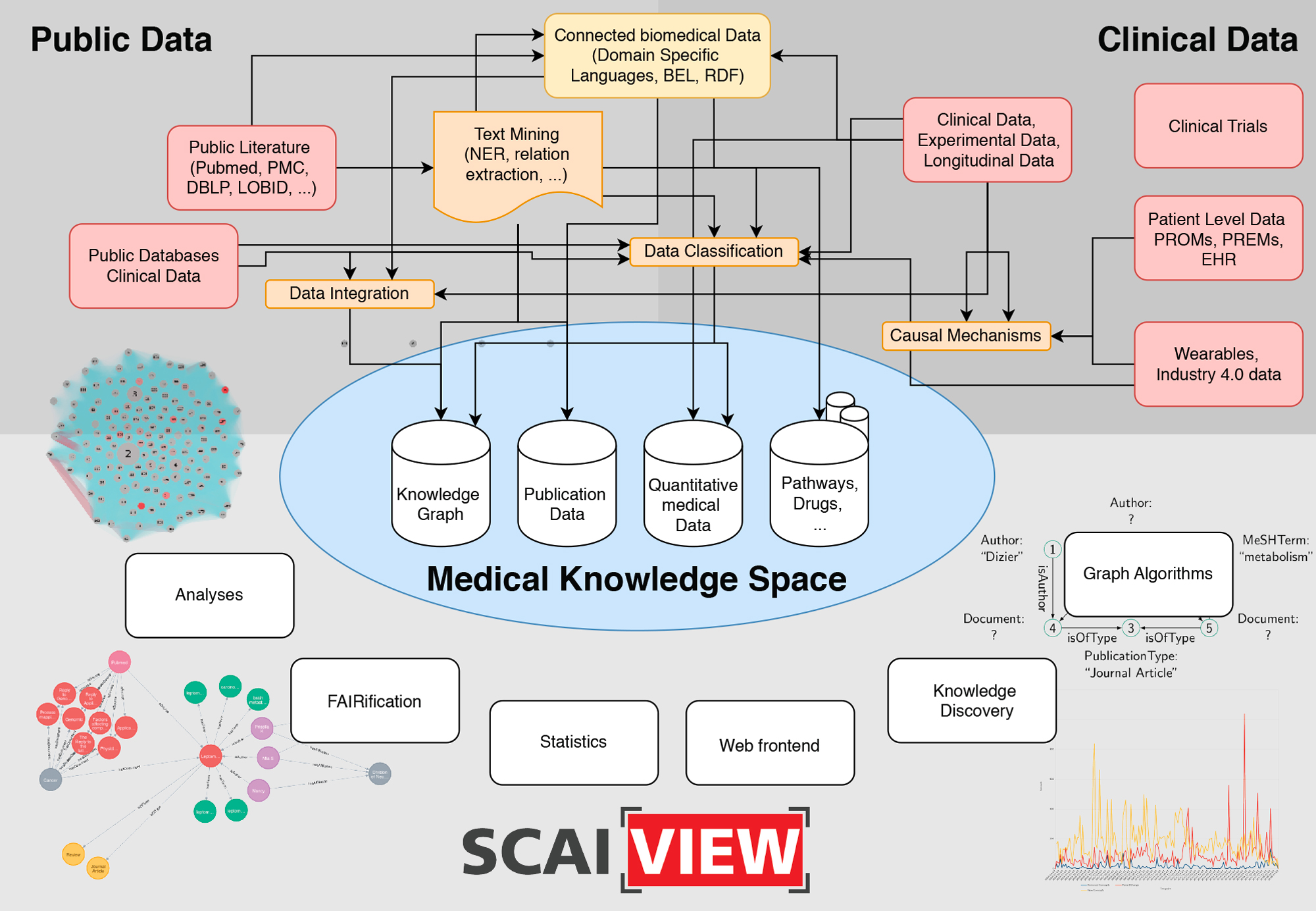
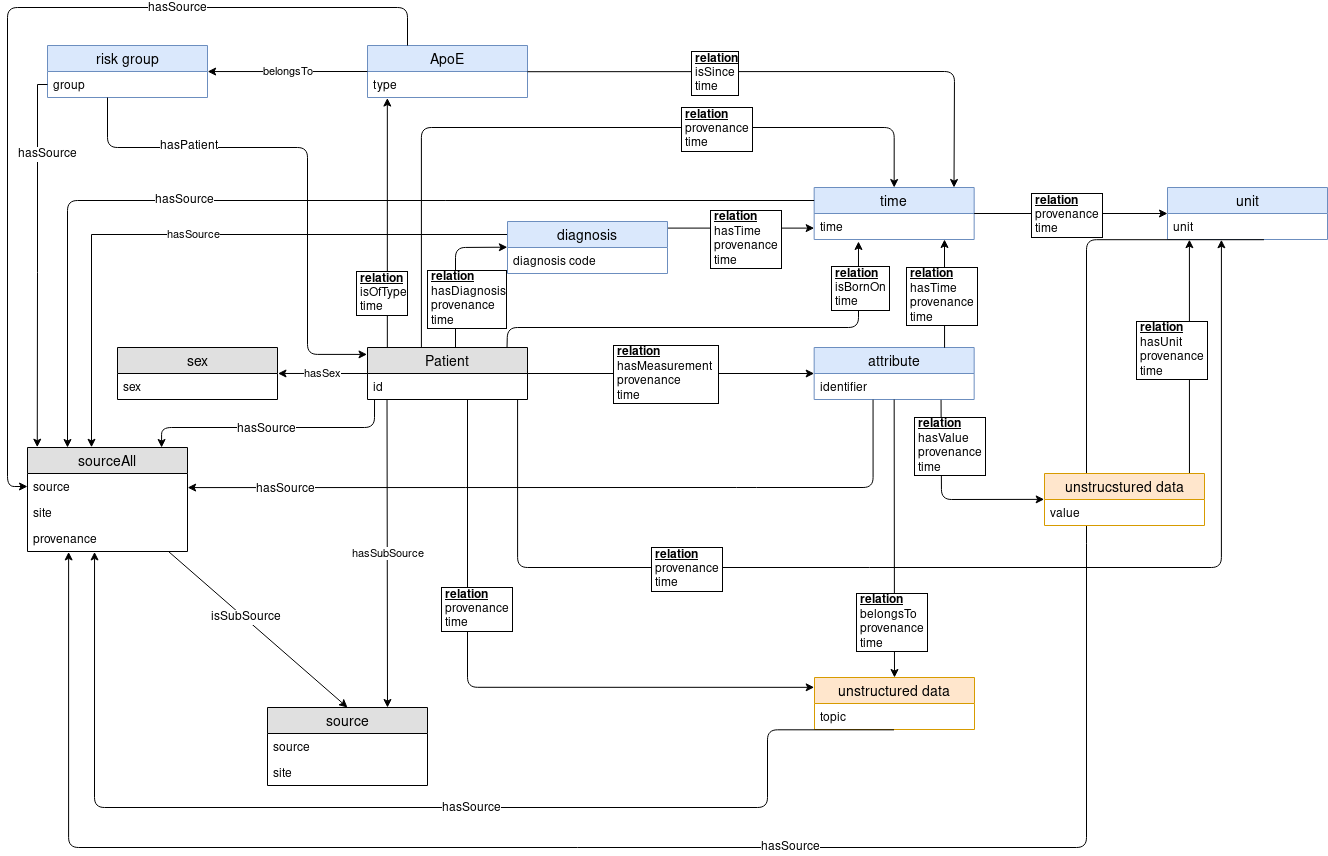
Der medizinische Wissensraum (Medical Knowledge Space) ist ein geeigneter Ort für klinische und medizinische Daten, sowohl aus öffentlichen Quellen als auch aus klinischen und patientenbezogenen Daten. Er bietet eine systematische Literatursuche und eine spezifische Analyse der verfügbaren klinischen Daten. Dies hilft, die relevantesten funktionalen Bereiche zu identifizieren, für die Big-Data-Konzepte entwickelt werden. Die detaillierte Analyse der Nutzerbedürfnisse sowie der beteiligten Daten kann durch eine eingehende Beratung und gegenseitige Lernfortschritte erfolgen, wobei alle verfügbaren Datenquellen in einer Interoperabilitätsschicht zusammengefasst werden.
Das Gesamtkonzept, das dem Medical Knowledge Space von SCAI.BIO zugrunde liegt, besteht darin, die Analyse des medizinischen Wissens patientenzentriert direkt zu den Daten oder Patienten zu bringen. Dabei ist es wichtig, indem wir Werkzeuge und Technologien entwickeln, die Patienten an jedem geografischen Ort erreichen können (Skalierbarkeit). Die enormen Fortschritte bei der Entwicklung von Geräten für die Gesundheitsfürsorge, einschließlich elektronischer Gesundheitsakten, Portaltechnologien und drahtloser Kommunikation, werden eine Schlüsselrolle in der künftigen Versorgung spielen. Von besonderem Interesse ist dabei beispielsweise auch, dass mobile Geräte für die Echtzeit-Überwachung eingesetzt werden können, einschließlich einer Vielzahl von Sensoren zur Erkennung von Veränderungen des Gesundheitszustands der Person.
Der Medical Knowledge Space bietet Methoden zur Harmonisierung und Interoperabilität, zur Vereinfachung ihrer leichten Auffindbarkeit, zur gemeinsamen Nutzung von Datenquellen sowie von Verarbeitungs-, Analyse- und Modellierungswerkzeugen und schließlich zu einer besseren Dokumentation und Reproduzierbarkeit. Bei der Entwicklung und anschließenden Bereitstellung der Umgebung geht man schrittweise vor. Dabei startet man mit in großen Stichproben gewonnenen Rohdaten (etwa Diagnosedaten, molekulare Daten oder klinische Informationen) und erarbeitet das das erforderliche Format für fortgeschrittene Datenanalyse und Modellierung. Hierbei ist die Interoperabilitätsschicht ein entscheidendes Bindeglied zwischen modellbasierten und klinischen Projekten. Wir stellen verschiedene Analysewerkzeuge und Schnittstellen zur Verfügung, um Graphenabfragen und Wissensentdeckung anzuwenden. Wann immer möglich, werden wir FAIR-Daten zur Verfügung stellen.
Literatur
[1] Dörpinghaus, J. and A. Stefan. "Optimization of Retrieval Algorithms on Large Scale Knowledge Graphs." arXiv preprint arXiv:2002.03686 (2020).
[2] Dörpinghaus, Jens, et al. "Towards context in large scale biomedical knowledge graphs." arXiv preprint arXiv:2001.08392 (2020).
[3] Dörpinghaus, Jens, Carsten Düing, and Vera Weil. "A Minimum Set-Cover Problem with several constraints." 2019 Federated Conference on Computer Science and Information Systems (FedCSIS). IEEE, 2019.
[4] Dörpinghaus, Jens, and Andreas Stefan. "Knowledge Extraction and Applications utilizing Context Data in Knowledge Graphs." 2019 Federated Conference on Computer Science and Information Systems (FedCSIS). IEEE, 2019.
[5] Dörpinghaus, Jens, and Marc Jacobs. "Semantic Knowledge Graph Embeddings for biomedical Research: Data Integration using Linked Open Data." Proceedings of the Posters and Demos Track of the 15th International Conference on Semantic Systems – SEMANTiCS2019, 2019.
[6] Dörpinghaus, J. et al. “Context graph for biomedical research data: A FAIR and open approach towards reproducible research in Medicine”, 3rd Annual MAQC Society Conference 2019.
[7] Dörpinghaus, J. et al. "SCAIView–A Semantic Search Engine for Biomedical Research Utilizing a Microservice Architecture." Proceedings of the Posters and Demos Track of the 14th International Conference on Semantic Systems – SEMANTiCS2018, 2018